全球千人基因组新突破:长读长测序 揭开人类DNA“暗物质”之谜

一项里程碑式的研究利用长读长测序技术揭示了人类 DNA 中大量以往未被检测到的结构变异,重塑了我们对遗传学和疾病潜力的理解。
最近发表在《Nature》期刊上的一项研究中,研究人员利用新一代“长读长”测序技术,探究了 DNA 中的大规模结构变异(SVs)——这些复杂的插入、缺失和重排现象目前尚不完全清楚。他们的突破性数据集涵盖了来自全球 26 个人群的 1,019 名个体。研究还采用了一种基于图形的创新分析框架,成功解析了超过 107,000 个双等位基因结构变异,并将这些数据公开共享。
这项高分辨率的基因组研究不仅显著深化了我们对人类遗传真实多样性的理解,还推动了我们对致病基因变异的识别和未来管理能力。

研究背景
生物学教科书通常将人类基因组描绘为由 30 亿个 A、T、G、C 组合而成的线性序列——这些 DNA 是我们生命的基石。然而,现实情况要复杂得多,DNA 中存在大规模的结构变异(SVs),包括整段 DNA 的缺失、重复、插入和倒位。
尽管这些结构变异占据了生物体之间大部分碱基对的差异,并且对人类健康具有重要影响,但它们的研究仍然非常困难且认知有限。目前主流的短读长测序技术将长 DNA 片段切割成微小片段后进行扩增。虽然这种方法对小变异有效,但在绘制复杂结构变异(尤其是大片段插入和多等位基因可变数目串联重复序列 VNTRs)时表现不佳,有时甚至会完全遗漏。
因此,人类基因组的大部分内容对科学和医学而言仍是未知领域,导致许多本可治愈的遗传疾病持续存在。长读长测序是一项相对新兴的技术,能够读取更长的连续 DNA 片段,从而克服短读长测序在结构变异研究中的主要缺陷。利用这项技术,我们有望揭示人类基因组中隐藏的部分及其蕴含的医学宝藏。
研究内容
本研究正是基于这一目标:一个跨国研究团队利用全球多样化队列开展了大规模的结构变异图谱绘制项目。研究样本来自“千人基因组计划”(1kGP),最初包括 1,064 个样本(淋巴母细胞系)。
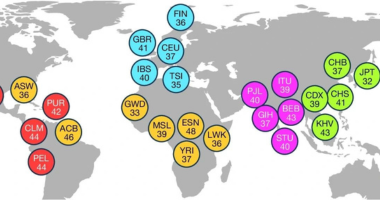
通过严格的质控流程(结合DNA浓度测定、纯度评估和片段长度验证),数据集最终缩减为 1,019 个样本,覆盖非洲、美洲、欧洲、东亚和南亚的 26 个不同 ancestry(祖先群体)。
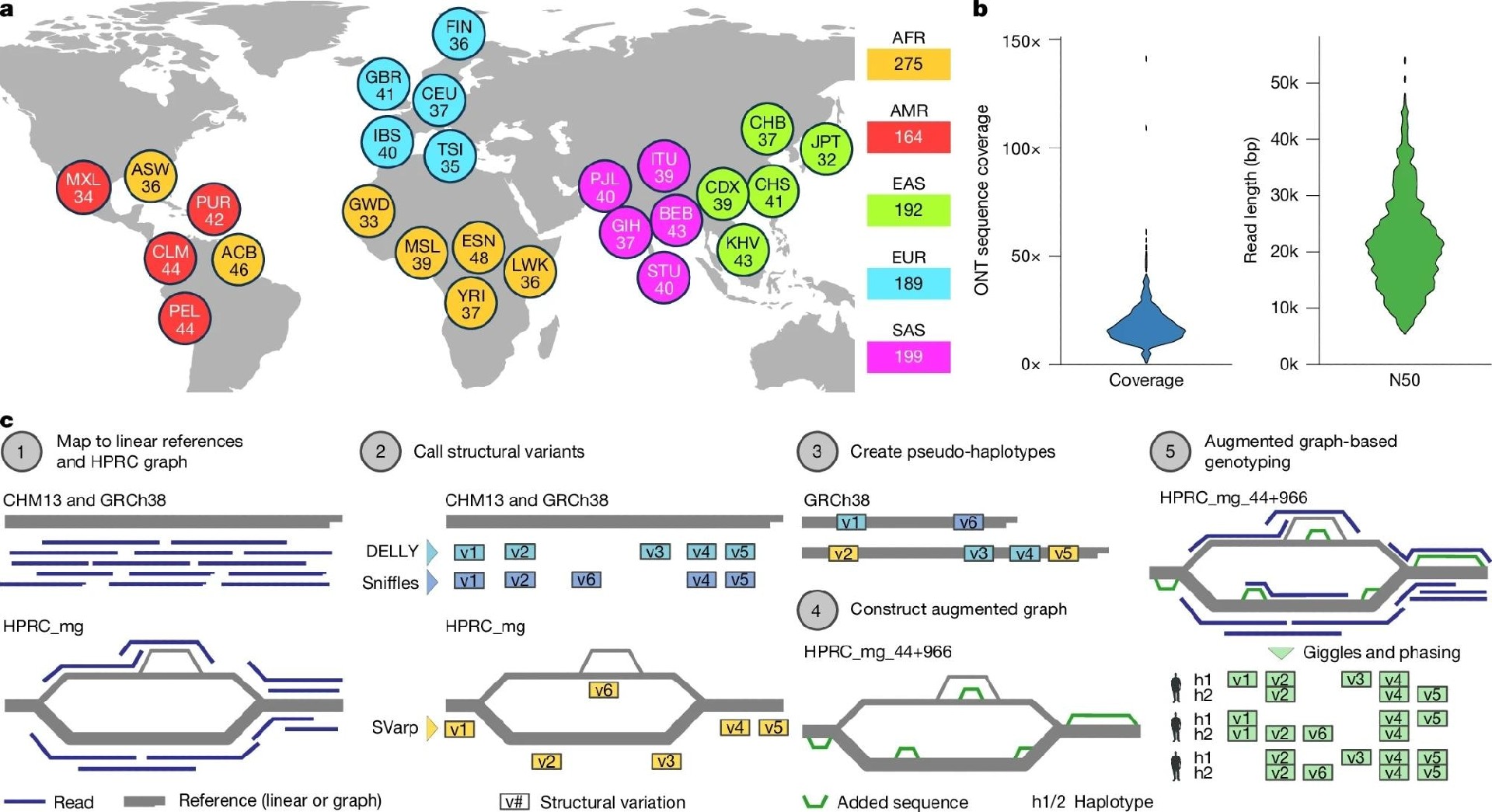
图:a. 1,019 个长读长基因组的自我认同地理祖先分析,代表了 5 个大陆地区的 26 个地理区域(即种群)。所使用的三字母代码等同于 1kGP 第三阶段中使用的代码 18 ,并在补充表 2 中解析。 b. 每个样本的ONT序列覆盖度,以倍数覆盖(左)和碱基对的 N50 读取长度(右)表示。 c. 使用泛基因组图增强方法进行图感知发现和 SV 基因分型的 SAGA 框架的示意图。
研究采用了牛津纳米孔技术(ONT)的长读长测序平台,该技术能够生成中位读长超过 20,000 个碱基对的数据。
为分析这一复杂数据集,团队开发了一种名为 SAGA(基于图形增强的结构变异分析)的新型计算框架。该框架包括四个关键步骤:1)将长读长序列对齐到线性和图形参考基因组;2)利用多种算法发现结构变异;3)通过图形增强整合新发现的结构变异;4)使用 Giggles 软件对队列进行基因分型。
研究发现
本研究成功创建了一个包含超过 10 万个双等位基因结构变异的公开目录,以及 369,685 个多等位基因 VNTRs。这些变异包括倒位、缺失、重复和插入,其中完全解析的插入位点数量增加了十倍以上,填补了人类基因组知识的关键空白。
通过家系三重样本验证,研究显示双等位基因结构变异的错误率极低(缺失和插入分别为 3.87% 和 4.44%)。值得注意的是,本研究中发现的新结构变异大多非常罕见,59.3% 的次要等位基因频率(MAF)低于 1%。非洲裔个体表现出最高程度的结构变异多样性。
此外,研究还揭示了结构变异形成的生物学机制,例如 L1 和 SVA 逆转录转座子通过启动子劫持等位点特异性过程促进结构变异的产生和易位。
研究意义
本研究标志着人类基因组学认知的重大飞跃。长读长测序技术的应用成功发现了更多结构变异(尤其是插入),而多样化样本队列(覆盖多个大洲的 26 个 ancestry)确保了研究结果的普适性和全球应用价值。
此外,这项全面且精确的结构变异图谱作为开放资源,为遗传医学开启了新时代,使我们能够识别和早期治疗以往未知的遗传疾病。在罕见病基因组分析中,该资源过滤了 55% 的候选变异,同时保留了 94%(35/37)的已验证致病变异。这一开放资源将为科学界提供宝贵支持,助力人类进化、群体遗传学和遗传变异功能后果的深入研究。
期刊参考
Schloissnig, S., Pani, S., Ebler, J., Hain, C., Tsapalou, V., Söylev, A., Hüther, P., Ashraf, H., Prodanov, T., Asparuhova, M., Magalhães, H., Höps, W., Sotelo-Fonseca, J. E., Fitzgerald, T., Santana-Garcia, W., Moreira-Pinhal, R., Hunt, S., Pérez-Llanos, F. J., Wollenweber, T. E., … Korbel, J. O. (2025). Structural variation in 1,019 diverse humans based on long-read sequencing. Nature. DOI – 10.1038/s41586-025-09290-7, https://www.nature.com/articles/s41586-025-09290-7
延展阅读
科学家发现超过5000种基因变异,使某些癌症能够茁壮成长
致命病毒冲破亚马逊!基因重配+人类活动引爆全球危机,首例胎儿死亡震惊世界